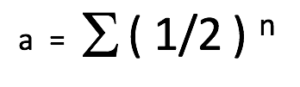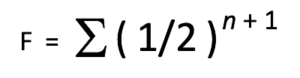Les pédigrées
 Les pédigrées sont les registres officiels de la filiation et donc de l’ascendance d’un animal. Ils font partie intégrante de la tenue des registres officiels requis par un club de race pour enregistrer de nouveaux animaux. Les pédigrées sont également des ressources précieuses pour la génétique d’un animal, grâce auxquelles il est possible d’estimer la contribution génétique d’un ancêtre particulier et de déterminer le lien génétique entre deux individus. Comme nous le verrons, le pédigrée est également utilisé pour déterminer les estimations de la consanguinité chez les animaux.
Les pédigrées sont les registres officiels de la filiation et donc de l’ascendance d’un animal. Ils font partie intégrante de la tenue des registres officiels requis par un club de race pour enregistrer de nouveaux animaux. Les pédigrées sont également des ressources précieuses pour la génétique d’un animal, grâce auxquelles il est possible d’estimer la contribution génétique d’un ancêtre particulier et de déterminer le lien génétique entre deux individus. Comme nous le verrons, le pédigrée est également utilisé pour déterminer les estimations de la consanguinité chez les animaux.
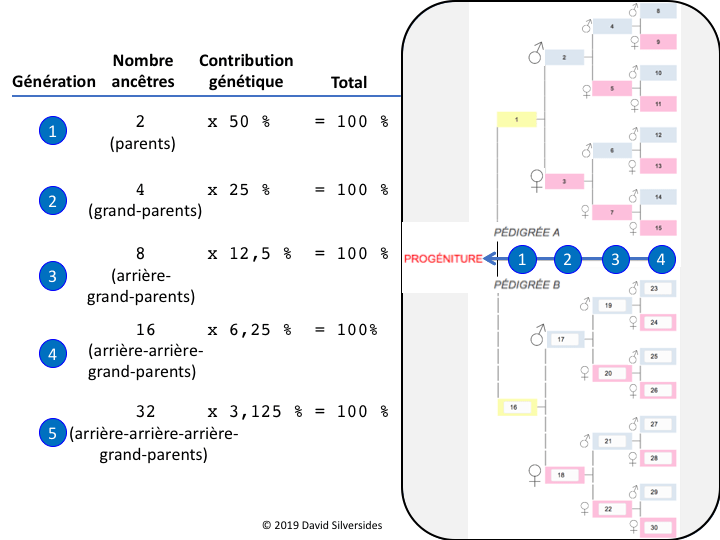
Les pédigrées sont organisés comme les branches d’un arbre : Dans le format standard, la contribution paternelle est présentée ci-dessus et la contribution maternelle est présentée ci-dessous pour l’animal (progéniture) en question. À chaque génération, la contribution génétique d’un ancêtre en particulier est divisée par deux. Ainsi, un animal (progéniture) a 2 parents dont chacun a contribué à 50% à la constitution génétique de leur progéniture et 4 grands-parents dont chacun a contribué à 25% à la constitution génétique de la progéniture, et ainsi de suite.
La consanguinité
Quand deux animaux avec des ancêtres communs sont accouplés ensemble, une condition de consanguinité existera dans la progéniture. La consanguinité a deux conséquences pour les phénotypes des animaux :
- Une augmentation de l’uniformité des phénotypes entre les animaux dans une génération avec une augmentation de « prépotence », c’est-à-dire la capacité de transmettre ou de « fixer » un phénotype d’une génération à la prochaine. Cette uniformité de phénotype est désirée par les éleveurs.
- La dépression de la consanguinité, qui inclue une diminution de la vitalité, un poids réduit, une réduction de la fertilité, un taux de croissance réduit, une augmentation des défauts congénitaux, une augmentation de mortalité, une espérance de vie plus courte que la normale et une augmentation des maladies génétiques de type récessif. La dépression de la consanguinité est cumulative : plus il y a de consanguinité, plus il y a de dépression de la consanguinité.
 Au niveau du génome, la consanguinité a pour effet d’augmenter le pourcentage des versions de gènes (allèles génétiques) homozygotes (N/N, M/M) et de diminuer les allèles hétérozygotes (porteurs ; M/N). Les mutations qui sont récessives vont s’accumuler dans l’homozygote (M/M), ce qui va augmenter la fréquence des maladies génétiques récessives qui vont contribuer à la dépression de la consanguinité.
Au niveau du génome, la consanguinité a pour effet d’augmenter le pourcentage des versions de gènes (allèles génétiques) homozygotes (N/N, M/M) et de diminuer les allèles hétérozygotes (porteurs ; M/N). Les mutations qui sont récessives vont s’accumuler dans l’homozygote (M/M), ce qui va augmenter la fréquence des maladies génétiques récessives qui vont contribuer à la dépression de la consanguinité.
Plus simplement, Dame Nature n’aime pas trop l’uniformité, ni en phénotype, ni en génotype. Elle aime beaucoup mieux les variations, la diversité et les différences.
Les races modernes de nos chats sont presque toutes par définition des groupes d’animaux consanguins. Le nombre limité des animaux fondateurs pour une race pure, les livres de généalogie fermés et la tendance à utiliser seulement une fraction des animaux d’une race pour produire la prochaine génération (« effet de champion » et « effet de mâle populaire ») font que nos races pures de chats sont composés d’animaux consanguins.
Le coefficient de relation (a) et le coefficient de consanguinité (COI)
En 1921-1922, Sewall Wright, un généticien et mathématicien américain, a défini deux valeurs mathématiques basées sur un pédigrée connu : le coefficient de relation (a) et le coefficient de consanguinité (COI ou parfois simplement « F »). Ces deux valeurs peuvent être utiles lorsqu’on veut prendre des décisions au sujet de l’accouplement des animaux domestiques. Ces coefficients ne sont pas des valeurs absolues et elles ne représentent qu’indirectement l’ADN et les gènes de l’animal. Ce sont des valeurs abstraites calculées, faites avec la supposition que tous nos gènes ont une hérédité mendélienne simple avec des allèles dominants et récessifs, ce qui est parfois simpliste. Même si ce sont des chiffres abstraits, le coefficient de relation et le coefficient de consanguinité sont des mesures utiles avec des applications pratiques. Ils permettent d’estimer la relation génétique entre deux animaux ainsi que la variation génétique dans le génome d’un animal en particulier. Ainsi, les éleveurs peuvent estimer les risques d’effets indésirables sur la santé (dépression de consanguinité) des générations futures en raison de l’absence de variation génétique due à trop de consanguinité.
Le coefficient de relation (a) est une estimation de la quantité d’ADN qui est partagée entre deux animaux dans un pédigrée. La formule le plus simple qui exprime ce coefficient est la suivante :
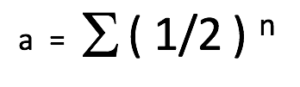
Où :
a = le coefficient de relation entre deux animaux
½ = la contribution génétique d’un parent vers son enfant
n = le nombre de chemins (ou nombre de générations) qui sépare les deux animaux dans le pédigrée. Autrement dit, le nombre de méioses (division cellulaire des cellules sexuelles) dans la lignée génétique ou le nombre de fois que le sexe productif s’est passé entre les deux animaux en question.
Σ = la somme des calculs pour toutes les voies possibles reliant les deux individus
Cette équation découle du fait que nous (et nos chats) sommes diploïdes et donc que nous avons reçu la moitié de notre l’ADN de notre mère et l’autre moitié de notre père. Autrement dit, l’ADN de nos parents a été divisé en deux (½) puis additionné (½ + ½ = 1,0) pour nous générer et créer notre propre ADN. Si deux animaux ne sont pas liés génétiquement, leur coefficient de relation est de 0. Au contraire, si deux animaux sont liés génétiquement (avec parents ou ancêtres communs) leur coefficient de relation sera supérieur à 0 et peut être calculé.
Le coefficient de consanguinité (COI, parfois dit « F ») est une estimation de la perte de variation génétique chez un animal donné, du fait de son ancêtre commun, tant du côté paternel que du côté maternel de son pedigree. Le coefficient de consanguinité peut être calculé à partir du coefficient de relation avec la formule suivante :
COI = (1/2) a = (1/2) n+1
Une présentation plus complète de la formule du coefficient de consanguinité est la suivante :
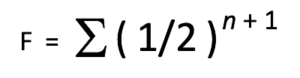
Où :
F = le coefficient de consanguinité (COI) pour l’individu en question.
½ = la contribution génétique d’un parent vers son enfant.
n = le nombre de chemins (ou nombre de générations) qui sépare les deux parents d’un individu à partir d’un ancêtre commun.
+1 = un facteur supplémentaire de (1/2) est ajouté pour représenter la perte moyenne de diversité génétique due à des ancêtres communs des côtés maternel et paternel de l’arbre généalogique.
∑ = la somme des calculs pour chaque ancêtre individuel en commun.
Si l’ancêtre en commun a lui-même des ancêtres en communs, alors il aura une valeur de coefficient de consanguinité positive. Le coefficient de consanguinité de l’animal en question est maintenant multiplié par le facteur de correction suivant:
(1 + Fa)
Où :
Fa = le coefficient de consanguinité de l’ancêtre en commun.
Pour les individus intrépides et les masochistes de la mathématique, les formules peuvent être plus complexe encore :
http://www.genetic-genealogy.co.uk/Toc115570144.html
http://www.genetic-genealogy.co.uk/Toc115570148.html.
Quelques exemples de coefficients de relation et de coefficients de consanguinité
Voici quelques exemples de coefficients de relation (la moyenne d’ADN commun) entre deux êtres apparentés qui n’ont aucun ancêtre commun. Le coefficient de consanguinité pour leur progéniture hypothétique est aussi calculé.
| Relation de parenté |
Coefficient de relation (a) |
Coefficient de consanguinité (COI)
de leur progéniture
|
| Parent – enfant |
50% |
25% |
| Frère – sœur |
50% |
25% |
| Grand-parent – petit-enfant |
25% |
12,5% |
| Oncle/tante – neveu/nièce |
25% |
12,5% |
| Demi-frère – demi-soeur |
25% |
12,5% |
| Cousin – cousine |
12,5% |
6,25% |
| Demi-cousins |
6,25% |
3,125% |
| Deuxième cousin |
3,13% |
1,063% |
Voici un exemple : un enfant et son parent partage 50 % de leur génétique, alors le coefficient de relation (a) est 50 %. L’enfant a un coefficient de consanguinité de 0. Un enfant issu du croisement entre un parent et son enfant ou entre un frère et une sœur aura un coefficient de consanguinité de 25%. Ça veut dire qu’en moyenne il y aura une perte de 25% de variation génétique (perte de M/N) et un gain de 25 % d’uniformité génétique (gain de N/N, M/M) au niveau de l’ADN de l’enfant. En ce qui concerne les humains, le mariage entre cousins est permis dans certains pays, même si leur descendance aura un coefficient de consanguinité de 6,25 %.
Consanguinité et la perte de diversité génétique
Encore une fois, pour un coefficient de consanguinité de 10%, cela signifierait que pour les sites génétiques hétérozygotes (M/N, porteurs) chez un ancêtre commun, il y a 10% de chance que ces locus génétiques deviennent homozygotes (N/N ou M/M) chez le descendant. Autrement dit, il y a en moyenne une perte nette de diversité génétique de 10% (ou un gain d’uniformité génétique si vous voulez) dans la progéniture en raison d’accouplements impliquant une consanguinité. Un locus génétique particulier qui a été converti d’hétérozygote (diversité) en homozygote (uniformité) chez une progéniture est engendré par la consanguinité dont celle-ci est dit identique par descendance due à l’ancêtre commun.
Les pédigrées
 Combien de générations d’un pédigrée doivent être prises en considération lorsqu’on calcule le coefficient de consanguinité ? La réponse est que plus il y a de générations, mieux c’est pour la validité du calcul. Utiliser un petit nombre de générations va donner une valeur de coefficient de consanguinité artificiellement basse comparé à la valeur obtenue lorsque plusieurs générations sont incluses. En pratique, lorsque l’on calcule le coefficient de consanguinité, toute l’information contenue dans le pédigrée connu est utilisée. Cette information peut être peut être limitée à trois générations ou elle peut contenir toutes les générations jusqu’aux animaux fondateurs de la race en question.
Combien de générations d’un pédigrée doivent être prises en considération lorsqu’on calcule le coefficient de consanguinité ? La réponse est que plus il y a de générations, mieux c’est pour la validité du calcul. Utiliser un petit nombre de générations va donner une valeur de coefficient de consanguinité artificiellement basse comparé à la valeur obtenue lorsque plusieurs générations sont incluses. En pratique, lorsque l’on calcule le coefficient de consanguinité, toute l’information contenue dans le pédigrée connu est utilisée. Cette information peut être peut être limitée à trois générations ou elle peut contenir toutes les générations jusqu’aux animaux fondateurs de la race en question.
Grace à la volonté des éleveurs et au pouvoir de l’internet, plusieurs pédigrées de nos chats sont disponibles sur les sites dédiés aux pédigrées. Un exemple d’un site pour les pédigrées des chats est PawPeds (https://pawpeds.com/).
Pour un animal donné, le pédigrée connu est présenté, les ancêtres communs sont notés, parfois le coefficient de consanguinité est calculé sur le nombre de générations connues. Souvent les éleveurs peuvent calculer et comparer les coefficients pour les accouplements futurs ou en d’autre mots, les accouplements virtuels.
Utilisation du coefficient de consanguinité
 Le coefficient de consanguinité, utilisé correctement, est un outil puissant pour les éleveurs quand vient le temps de choisir les parents pour les futures portées. De façon générale, un coefficient de consanguinité est une indication de l’état de santé global du génome d’un animal. Le génome est la somme totale de tous les gènes qui sont nécessaire pour la formation et la fonction de l’animal en question. Plus spécifiquement, un coefficient de consanguinité représente un pourcentage numérique basé sur une analyse de pédigrée qui reflète la perte de la variation génétique dont celle-ci se produit s’il y a des ancêtres communs sur les côtés paternel et maternel du pédigrée chez un individu, comme il a été mentionné plus haut. Avoir des ancêtres communs sur les deux côtés du pédigrée aura pour résultat d’obtenir des sites génétiques homozygotes (soit N/N ou M/M) chez les descendant à la place des sites hétérozygotes (M/N) chez les ancêtres porteurs. Cette condition est dite identique par descendance du fait d’avoir un ancêtre commun.
Le coefficient de consanguinité, utilisé correctement, est un outil puissant pour les éleveurs quand vient le temps de choisir les parents pour les futures portées. De façon générale, un coefficient de consanguinité est une indication de l’état de santé global du génome d’un animal. Le génome est la somme totale de tous les gènes qui sont nécessaire pour la formation et la fonction de l’animal en question. Plus spécifiquement, un coefficient de consanguinité représente un pourcentage numérique basé sur une analyse de pédigrée qui reflète la perte de la variation génétique dont celle-ci se produit s’il y a des ancêtres communs sur les côtés paternel et maternel du pédigrée chez un individu, comme il a été mentionné plus haut. Avoir des ancêtres communs sur les deux côtés du pédigrée aura pour résultat d’obtenir des sites génétiques homozygotes (soit N/N ou M/M) chez les descendant à la place des sites hétérozygotes (M/N) chez les ancêtres porteurs. Cette condition est dite identique par descendance du fait d’avoir un ancêtre commun.
Du côté génétique, une perte de variation génétique n’est pas désirable car il en résulte de la « dépression de consanguinité ». La dépression de consanguinité est bien documentée chez plusieurs espèces, autant pour les animaux que pour les plantes. L’effet de la reproduction « outbreeding » ou « crossbreeding » est aussi bien documenté. Celui-ci va augmenter la variation génétique du génome d’un animal pour donner la « vigueur hybride », aussi dit l’hétérose.
 Il est important de garder en tête que le coefficient de consanguinité ne représente pas le statut de variation pour les gènes spécifiques, mais plutôt le statut de variation global et estimé pour le génome au complet. S’il n’y a pas d’ancêtres communs entre le côté paternel et le côté maternel d’un pédigrée, la progéniture n’aura aucune perte de variation génétique par rapport à une population standard. La progéniture aura un coefficient de consanguinité de 0. S’il y a des ancêtres communs entre le côté paternel et le côté maternel d’un pédigrée, il y a maintenant un potentiel de perte de variation génétique dans le génome de la progéniture (identique par descendance) qui peut amener à la dépression de la consanguinité.
Il est important de garder en tête que le coefficient de consanguinité ne représente pas le statut de variation pour les gènes spécifiques, mais plutôt le statut de variation global et estimé pour le génome au complet. S’il n’y a pas d’ancêtres communs entre le côté paternel et le côté maternel d’un pédigrée, la progéniture n’aura aucune perte de variation génétique par rapport à une population standard. La progéniture aura un coefficient de consanguinité de 0. S’il y a des ancêtres communs entre le côté paternel et le côté maternel d’un pédigrée, il y a maintenant un potentiel de perte de variation génétique dans le génome de la progéniture (identique par descendance) qui peut amener à la dépression de la consanguinité.
Le coefficient de consanguinité est maintenant un chiffre supérieur à 0, souvent exprimé en pourcentage. Il est estimé que quand la consanguinité se produit, pour chaque 1% d’augmentation dans le coefficient de consanguinité, il y a une réduction de 1 % dans les traits mesurés (dépression de consanguinité). Le coefficient de consanguinité est le plus utile pour estimer les effets de consanguinité récents.
Il n’est pas surprenant que Dame Nature aime la variation génétique, car c’est la clé à long terme pour la survie et l’évolution d’une espèce. Malheureusement, pour nos animaux domestiques, les standards de races favorisent l’élevage pour l’uniformité du phénotype (élever pour « type »). Ceci implique généralement un certain niveau de consanguinité avec l’uniformité du génotype et une variation génétique réduite comme résultat.
L’interprétation du coefficient de la consanguinité
 Une valeur de coefficient de consanguinité élevée augmentera les risques d’expression des effets nuisibles de la consanguinité (dépression de la consanguinité). Par contre, ce même coefficient de consanguinité élevé permet aussi d’augmenter les chances que les traits désirables en lien avec la race en question soient fixés. Ainsi, le coefficient de consanguinité peut être vu comme un compromis. Les effets délétères associés à la consanguinité commencent à être observés lorsque le coefficient de consanguinité est supérieur à 5 %, c’est-à-dire juste un peu en dessous de la valeur du coefficient de consanguinité d’un croisement entre deux cousins (=6,25 %).
Une valeur de coefficient de consanguinité élevée augmentera les risques d’expression des effets nuisibles de la consanguinité (dépression de la consanguinité). Par contre, ce même coefficient de consanguinité élevé permet aussi d’augmenter les chances que les traits désirables en lien avec la race en question soient fixés. Ainsi, le coefficient de consanguinité peut être vu comme un compromis. Les effets délétères associés à la consanguinité commencent à être observés lorsque le coefficient de consanguinité est supérieur à 5 %, c’est-à-dire juste un peu en dessous de la valeur du coefficient de consanguinité d’un croisement entre deux cousins (=6,25 %).
Il est conseillé de maintenir un coefficient de consanguinité inférieur à 10 %, ce qui devrait permettre de garder stable (fixer) un certain nombre de traits désirables sans toutefois que les effets nuisibles de la consanguinité soient trop prononcés. Ainsi, il faut éliminer les croisements incestueux, avec un coefficient de consanguinité plus élevé que 12,5 %, par exemple les accouplements entre un parent et son enfant, entre un frère et une sœur, entre un grand-parent et son petit-enfant et entre un demi-frère et une demi-sœur. En pratique, il est recommandé de choisir un croisement qui va donner une valeur de coefficient de consanguinité qui est inférieure au coefficient de consanguinité moyen de la race en question (si c’est connu). Si plusieurs possibilités de croisements qui donnent un coefficient inférieur à la moyenne sont disponibles, il faut idéalement choisir celui qui donne le coefficient de consanguinité le plus bas, toujours en gardant les traits désirés de la race.